- 0
- 0
- 0
分享
- Meta打造首个「蛋白质宇宙」全景图!用150亿参数语言模型,预测了6亿+蛋白质结构
-
2022-11-02
新智元报道

新智元报道
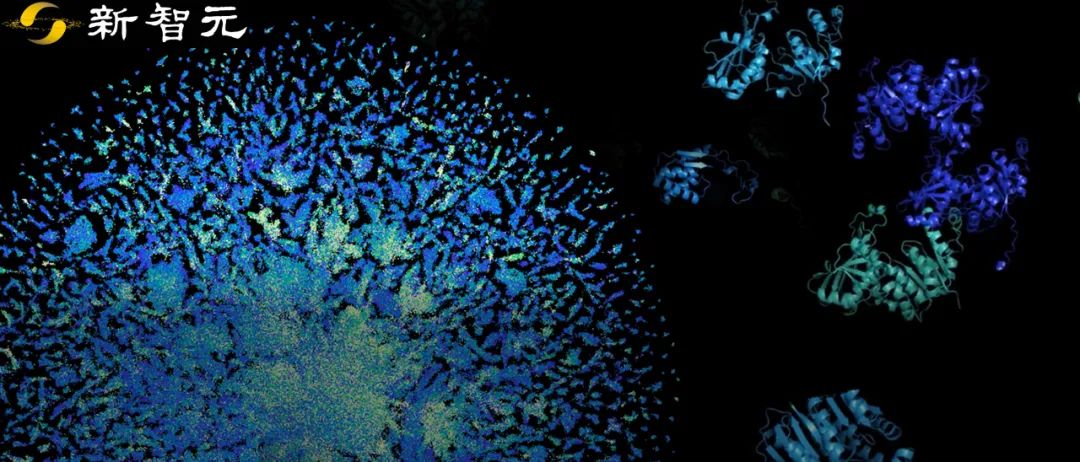
【新智元导读】Meta AI在蛋白质结构预测上又有新突破!利用大型语言模型 ,打造了一个超过6亿个宏基因组结构的数据库,让蛋白质结构预测提速60倍。
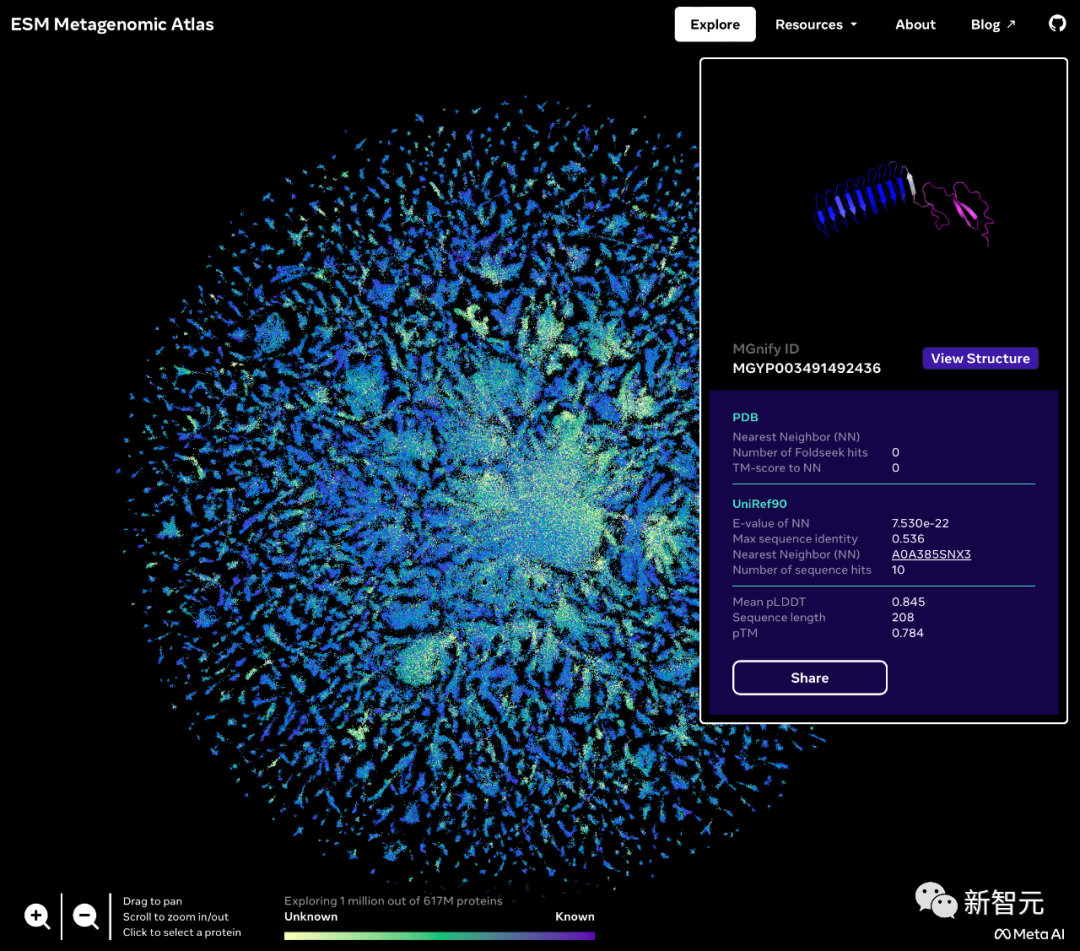
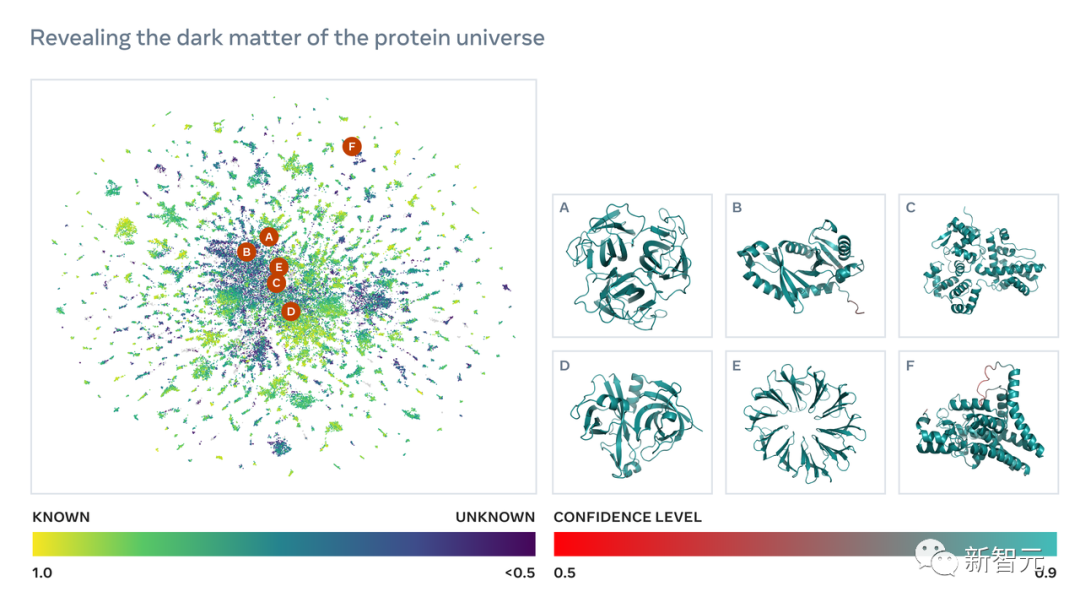
蛋白质宇宙的「暗物质」

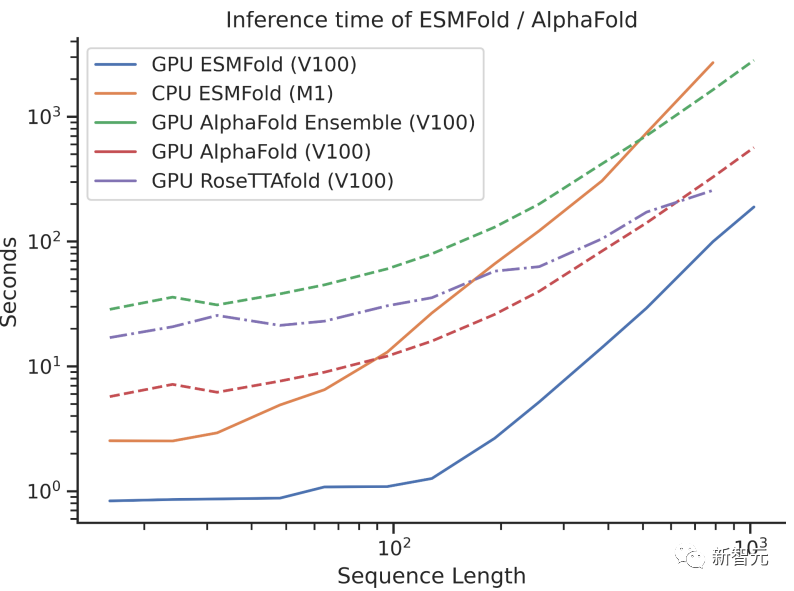
蛋白质结构预测,提速60倍!

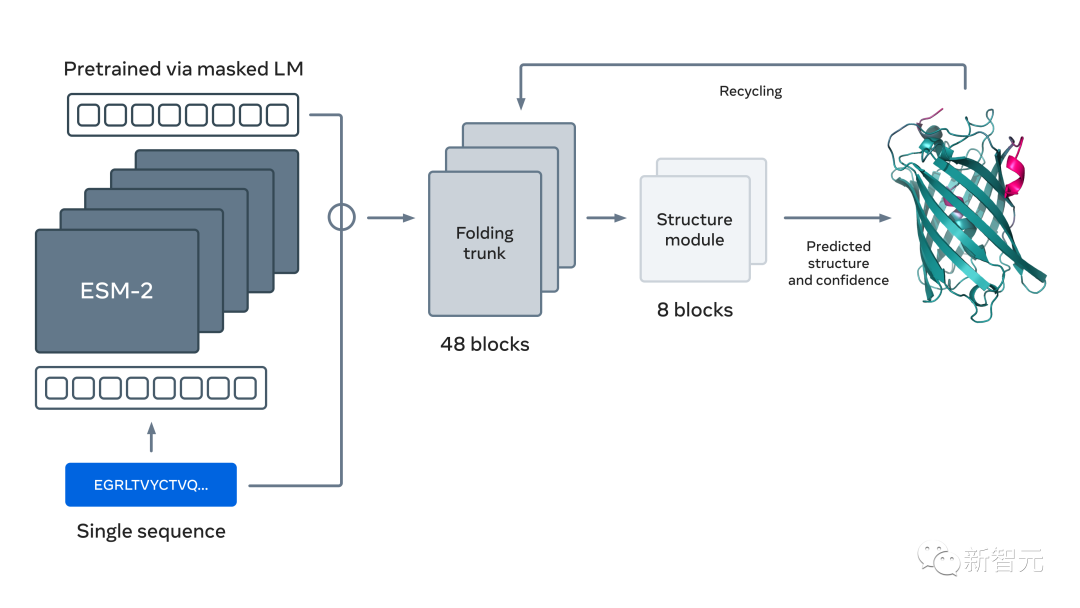
语言模型,真是「万能」的
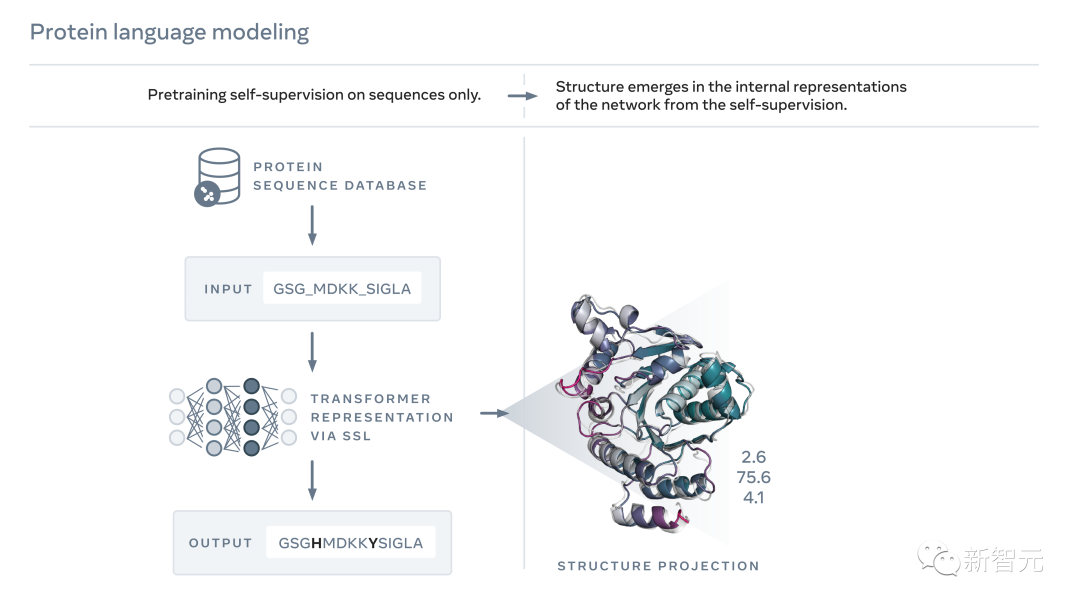


读懂「蛋白质语言」,让生命更透明


-
阅读原文
* 文章为作者独立观点,不代表数艺网立场转载须知
- 本文内容由数艺网收录采集自微信公众号新智元 ,并经数艺网进行了排版优化。转载此文章请在文章开头和结尾标注“作者”、“来源:数艺网” 并附上本页链接: 如您不希望被数艺网所收录,感觉到侵犯到了您的权益,请及时告知数艺网,我们表示诚挚的歉意,并及时处理或删除。













